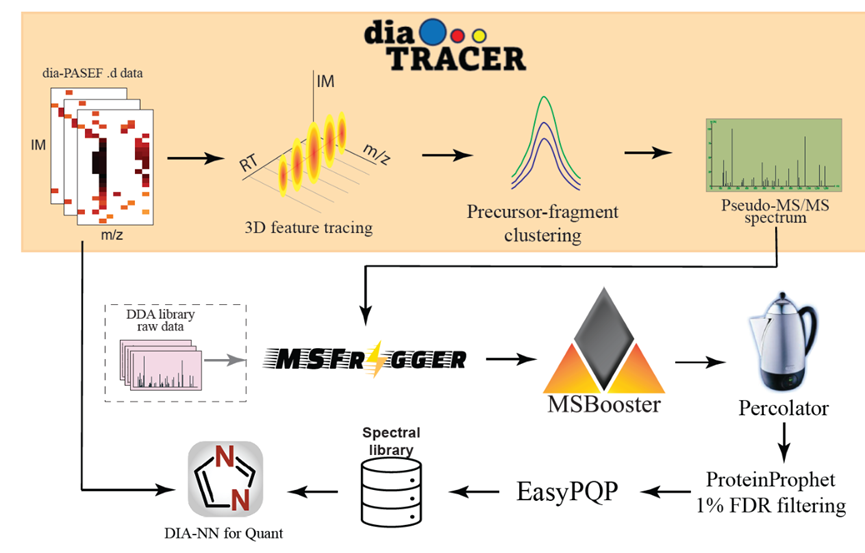
来自密歇根大学的Alexey I. Nesvizhskii团队日前在BioRxiv上公开了他们开发的新工具diaTracer,专门用于分析diaPASEF技术生成的蛋白质组学数据。论文标题:diaTracer enables spectrum-centric analysis of diaPASEF proteomics data;链接:https://doi.org/10.1101/2024.05.25.595875。
diaPASEF通过整合离子迁移分离(PASEF)技术在DIA基础上实现了更高的定量准确性和组学检测深度。diaTracer通过在质谱文件种识别基于质荷比、保留时间、以及离子迁移率形成的三维峰来查找并生成“假”的MS/MS谱图,生成的mzML文件可以以DDA-MS的方式进行后续的分析——没错,diaTracer就是DIA-Umpire的timsTOF特供版。
论文展示了diaTracer在处理脑脊液和血浆样本数据、磷酸化蛋白质组学和HLA免疫肽组学实验数据,以及痕量空间蛋白质组学研究数据方面的一系列表现。由于这种信号提取理论上无偏倚,因此diaTracer能够通过开放搜索模式、或质量偏移搜索模式,实现无限制地识别diaPASEF数据中的各种翻译后修饰。Data miner们狂喜。
DIA-Umpire的实际应用效果虽然见仁见智,但其缩小质谱搜索空间(search space)的想法在其发表于Nature Methods的2015年可谓开创之举。现在有了diaTracer、而且集成在了蛋白质组分析软件“香饽饽”FragPipe上(目前只支持命令行操作),spectrum-centric, library-free的分析范式会不会取代AI-assisted predicted library-based data search(毕竟还是用到了外源生成的库,因此笔者仍建议把这种分析范式归类为peptide-centric, library-based)成为新潮流,我们拭目以待。